Klassifizierung von Ameisen mithilfe von Künstlicher Intelligenz (KI)
Ameisen sind eines der vielfältigsten Insekten auf unserem Planeten. Derzeit gibt es etwa 16.000 bestätigte Ameisenarten, aber viele weitere gilt es noch zu entdecken und taxonomisch zu beschreiben[1]. In Anbetracht des extremen Zeitaufwandes, der sich für Wissenschaftler bei der taxonomischen Bestimmung ergibt, ist es naheliegend, nach alternativen Methoden zu suchen. Ziel unserer Arbeit war es daher zu testen, inwieweit sich die weitgehend optisch basierte Arbeit der Wissenschaftler mit Hilfe von bilderkennender Künstlicher Intelligenz (KI) unterstützen lässt.
Ameisen werden in ökologischen Studien häufig beispielhaft für alle anderen Tierarten, v.a. Insekten, verwendet. Das liegt zum einen daran, dass Ameisen sehr zahlreich in nahezu allen Ökosystemen des Planeten vorkommen, und zum anderen, dass ihre Artenvielfalt vergleichsweise gut erforscht ist. Letzteres gilt allerdings nicht für die Tropen; hier ist je nach Gebiet immer noch weit über die Hälfte aller Arten unbekannt.
Für das Projekt “Ecological and Socioeconomic Functions of Tropical Lowland Rainforest Transformation Systems” (EFForTS)[2] – ansässig an der Georg-August-Universität Göttingen – sammelten Forscher im Rahmen einer groß angelegten ökologischen Studie fast 135.000 Ameisen aus vier verschiedenen Agrar-Systemen im Tiefland der Provinz Jambi auf Sumatra in Indonesien. Ein Team von geschulten Wissenschaftlern und Studenten bestimmte diese Vielzahl an Individuen auf 336 Ameisenarten, die sich auf 74 Gattungen aus 9 Unterfamilien der Ameisen verteilen[3]. Jedoch war diese Sortierung der Proben äußerst zeitaufwändig: Die Forscher benötigten beinahe fünf Jahre für eine erste komplette Bestimmung aller Individuen und ein weiteres Jahr für eine Revision unter Einbeziehung externer taxonomischer Experten.
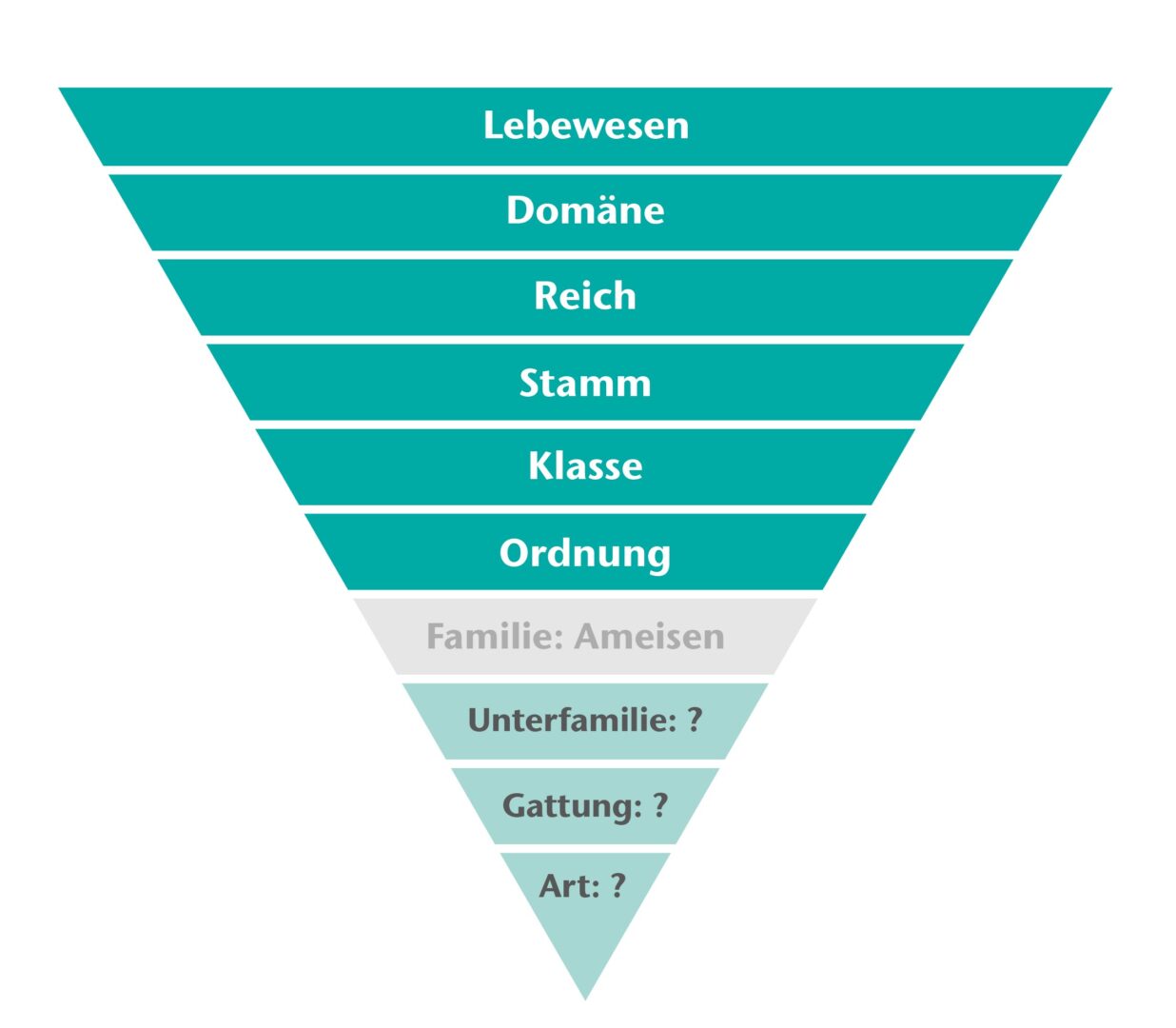
Unser Ziel besteht daher darin, die Arbeit der Biologen durch die Verwendung von Künstlicher Intelligenz (KI) für die Vorhersage der taxonomischen Hierarchie von Ameisen (Unterfamilie – Gattung – Art, s. Abb. 1) zu erleichtern.
Für jede Ebene der taxonomischen Hierarchie wurde ein eigenes KI-Modell erstellt, wobei die frontal aufgenommenen Ameisenbilder (Kopfperspektive) aus der AntWeb-Datenbank als Trainings-Datensatz verwendet wurde.
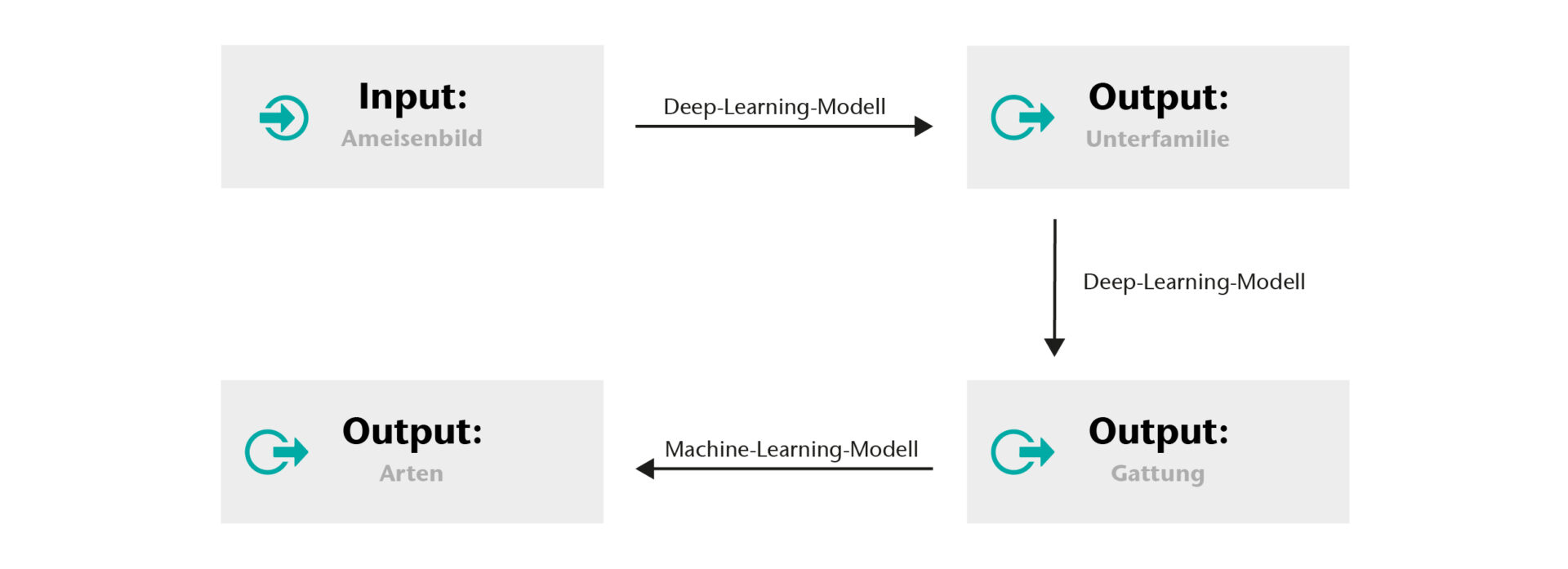
Zur Nutzung der KI-Modelle wurde ein Web-Prototyp entwickelt: Der Benutzer lädt ein unbekanntes Ameisenbild in die Oberfläche und erhält als Ergebnis eine Einschätzung zur taxonomischen Hierarchie mit einer angegebenen Vorhersagewahrscheinlichkeit. Der komplette Workflow ist in Abbildung 2 dargestellt.
Für jede Ebene der taxonomischen Hierarchie wurde ein eigenes Modell erstellt, um auf jeder Ebene möglichst zuverlässige Ergebnisse zu erhalten. Wenn für ein Eingabebild die maximale Vorhersagewahrscheinlichkeit für die Unterfamilienebene größer als 90% ist, wird dem Benutzer nur die Unterfamilie mit dieser höchsten Wahrscheinlichkeit angezeigt. In anderen Fällen wird eine Rangfolge der ersten drei vorhergesagten Unterfamilien mit der höchsten Vorhersagewahrscheinlichkeit dargestellt.
Das Deep-Learning-Modell mit Inception V3-Architektur für die Vorhersage von Unterfamilien wurde an Kopfbildern trainiert, die zu den folgenden Unterfamilien gehören: Amblyoponinae, Dolichoderinae, Dorylinae, Formicinae, Myrmicinae und Ponerinae.
Die Klassifizierung auf Gattungsebene erfolgte ebenfalls mit einem Deep-Learning-Modell mit Inception V3-Modellarchitektur. Für jede Unterfamilie wurde ein eigenes Modell auf Gattungen dieser Unterfamilie trainiert. Die Klassifizierung auf Gattungsebene war für die Unterfamilien Amblyoponinae, Dolichoderinae, Dorylinae, Ectatomminae, Formicinae, Myrmicinae Ponerinae, Proceratiinae und Pseudomyrmecinae möglich.
Um eine Vorhersage der Art zu ermöglichen, wurden Gattungen mit mindestens zwei Arten ausgewählt, wobei die verfügbare Anzahl der Kopfbilder in jeder Art 10 oder mehr beträgt. Für die ausgewählten Gattungen wurde ein maschinelles Lernmodell mit der Art als Klasse (Label) erstellt.
Aktuell arbeiten wir gemeinsam mit den Wissenschaftlern an der Veröffentlichung eines “Methodenpapiers” in einer von Fachkollegen begutachteten wissenschaftlichen Zeitschrift, die sich auf die Förderung neuer Methoden in Ökologie und Evolution konzentriert.
[1] Nazarreta R, Buchori D, Hidayat P, Fardiansiah R, Scheu S, Drescher J. 2019. A Guide to the Ants of Jambi (Sumatra, Indonesia) –Identification key to Common Ant Genera and Images of the EFForTS collection. Version 4.0. Animal Ecology, Johann-Friedrich-Blumenbach Institute for Zoology and Anthropology, University of Goettingen, Germany
[2] Drescher J & Rembold K, Allen K, Beckschäfer P, Buchoiori D, Clough Y, Faust H, Fauzi AM, Gunawan D, Hertel D, Irawan B, Jaya INS, Klarber B, Kleinn C, Knohl C, Kotowska MM, Krashevska V, Krishna V, Leuschner C, Lorenz W, Meijide A, Melati D, Nomura M, Rérez-Cruzado C, Qaim M, Siregar IZ, Steinebach S, Tjoa A, Tscharntke T, Wick B, Wiegand K, Kreft H, Scheu S (2016). Ecological and socio-economic functions across tropical land use systems after rainforert conversion. Philosophical Transactions of the Royal Society B 371: 20150275
[3] https://www.uni-goettingen.de/de/a+guide+to+the+ants+of+jambi+%28sumatra%2c+indonesia%29+-+identification+key+to+common+ant+genera+and+images+of+the+efforts+collection+%28z02%29/609422.html
War dieser Artikel hilfreich für Sie? Oder haben Sie Fragen? Schreiben Sie uns einfach einen Kommentar oder rufen Sie uns gerne an.
Über den Autor
Ulrike Pick leitet bei der CONET Solutions GmbH den Lösungsbereich Data Intelligence. Die staatlich geprüfte Informatikerin verfügt über umfangreiche Erfahrung in Anwendungsentwicklung, Lösungskonzeption und Projektleitung in den Bereichen Datenanalyse und Informationsqualität, Business Analytics und Big Data.